Mapeo de genomas
El mapeo del genoma es el proceso de encontrar la ubicación de los genes en cada cromosoma. Los mapas que se crean son comparables a los mapas que utilizamos para navegar por las calles. Un mapa genético es una ilustración que enumera los genes y su ubicación en un cromosoma. Los mapas genéticos proporcionan una imagen general (similar a un mapa de carreteras interestatales) y usan marcadores genéticos (similares a puntos de referencia). Un marcador genético es un gen o secuencia en un cromosoma que muestra un enlace genético con un rasgo de interés. El marcador genético tiende a heredarse con el gen de interés, y una medida de la distancia entre ellos es la frecuencia de recombinación durante la meiosis. Los primeros genetistas llamaron a este análisis de vinculación.
Los mapas físicos entran en los detalles íntimos de las regiones más pequeñas de los cromosomas (similar a un mapa de ruta detallado). Un mapa físico es una representación de la distancia física, en nucleótidos, entre genes o marcadores genéticos. Se requieren tanto mapas de enlace genético como mapas físicos para construir una imagen completa del genoma. Tener un mapa completo del genoma facilita a los investigadores el estudio de genes individuales. Los mapas del genoma humano ayudan a los investigadores en sus esfuerzos por identificar genes humanos que causan enfermedades relacionadas con enfermedades como el cáncer, enfermedades cardíacas y fibrosis quística, por nombrar algunos. Además, el mapeo del genoma se puede utilizar para ayudar a identificar organismos con rasgos beneficiosos, como los microbios con la capacidad de limpiar contaminantes o incluso prevenir la contaminación. La investigación que involucra el mapeo del genoma de la planta puede conducir a métodos que produzcan mayores rendimientos de cultivos o al desarrollo de plantas que se adapten mejor al cambio climático.
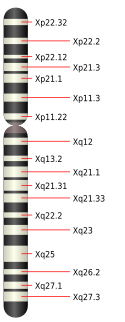 |
| Este es un mapa físico del cromosoma X humano. |
Secuenciación del genoma completo
Aunque ha habido avances significativos en las ciencias médicas en los últimos años, los médicos todavía están confundidos por muchas enfermedades y los investigadores están utilizando la secuenciación del genoma completo para llegar al fondo del problema. La secuenciación del genoma completo es un proceso que determina la secuencia de ADN de un genoma completo. La secuenciación del genoma completo es un enfoque de fuerza bruta para la resolución de problemas cuando existe una base genética en el núcleo de una enfermedad. Varios laboratorios ahora brindan servicios para secuenciar, analizar e interpretar genomas completos.
En 2010, se utilizó la secuenciación completa del genoma para salvar a un niño cuyos intestinos tenían múltiples abscesos misteriosos. El niño tuvo varias operaciones de colon sin alivio. Finalmente, una secuencia completa del genoma reveló un defecto en una vía que controla la apoptosis (muerte celular programada). Se utilizó un trasplante de médula ósea para superar este trastorno genético, lo que condujo a una cura para el niño. Fue la primera persona en ser diagnosticada con éxito utilizando la secuenciación del genoma completo.
Los primeros genomas secuenciados, como los que pertenecen a virus, bacterias y levaduras, fueron más pequeños en términos de cantidad de nucleótidos que los genomas de organismos multicelulares. Ahora se conocen los genomas de otros organismos modelo, como el ratón (Mus musculus), la mosca de la fruta (Drosophila melanogaster) y el nematodo (Caenorhabditis elegans). Se realiza una gran cantidad de investigación básica en organismos modelo porque la información se puede aplicar a otros organismos. Un organismo modelo es una especie que se estudia como modelo para comprender los procesos biológicos en otras especies que pueden ser representados por el organismo modelo. Por ejemplo, las moscas de la fruta pueden metabolizar el alcohol como los humanos, por lo que los genes que afectan la sensibilidad al alcohol se han estudiado en las moscas de la fruta en un esfuerzo por comprender la variación en la sensibilidad al alcohol en los humanos. Tener genomas completos secuenciados ayuda con los esfuerzos de investigación en estos organismos modelo.
| Se realiza mucha investigación básica con organismos modelo, como el ratón, Mus musculus; la mosca de la fruta, Drosophila melanogaster; el nematodo Caenorhabditis elegans; la levadura Saccharomyces cerevisiae; y la hierba común, Arabidopsis thaliana. |
La primera secuencia del genoma humano se publicó en 2003. El número de genomas completos que se han secuenciado aumenta constantemente y ahora incluye cientos de especies y miles de genomas humanos individuales.
Aplicando Genómica
La introducción de la secuencia de ADN y los proyectos de secuenciación del genoma completo, particularmente el Proyecto del Genoma Humano, ha ampliado la aplicabilidad de la información de la secuencia de ADN. La genómica ahora se está utilizando en una amplia variedad de campos, como la metagenómica, la farmacogenómica y la genómica mitocondrial. La aplicación más conocida de la genómica es comprender y encontrar curas para las enfermedades.
Predecir el riesgo de enfermedad a nivel individual
La predicción del riesgo de enfermedad implica la detección e identificación de individuos actualmente sanos por análisis del genoma a nivel individual. Se puede recomendar la intervención con cambios en el estilo de vida y medicamentos antes del inicio de la enfermedad. Sin embargo, este enfoque es más aplicable cuando el problema surge de una sola mutación genética. Dichos defectos solo representan alrededor del 5 por ciento de las enfermedades que se encuentran en los países desarrollados. La mayoría de las enfermedades comunes, como la enfermedad cardíaca, son multifactoriales o poligénicas, lo que se refiere a una característica fenotípica que está determinada por dos o más genes, y también a factores ambientales como la dieta. En abril de 2010, los científicos de la Universidad de Stanford publicaron el análisis del genoma de un individuo sano (Stephen Quake, científico de la Universidad de Stanford, a quien le secuenciaron el genoma); El análisis predijo su propensión a adquirir diversas enfermedades. Se realizó una evaluación de riesgos para analizar el porcentaje de riesgo de Quake para 55 afecciones médicas diferentes. Se encontró una rara mutación genética que mostró que estaba en riesgo de sufrir un ataque cardíaco repentino. También se predijo que tenía un riesgo del 23 por ciento de desarrollar cáncer de próstata y un riesgo del 1,4 por ciento de desarrollar la enfermedad de Alzheimer. Los científicos utilizaron bases de datos y varias publicaciones para analizar los datos genómicos. Si bien la secuenciación genómica se está volviendo más asequible y las herramientas analíticas se están volviendo más confiables, quedan por abordar los problemas éticos que rodean el análisis genómico a nivel de la población. Por ejemplo, ¿podrían esos datos utilizarse legítimamente para cobrar más o menos por el seguro o para afectar las calificaciones crediticias?
Estudios de asociación de genoma completo
Desde 2005, ha sido posible realizar un tipo de estudio llamado estudio de asociación de todo el genoma, o GWAS. Un GWAS es un método que identifica diferencias entre individuos en polimorfismos de un solo nucleótido (SNP) que pueden estar involucrados en causar enfermedades. El método es particularmente adecuado para enfermedades que pueden verse afectadas por uno o muchos cambios genéticos en todo el genoma. Es muy difícil identificar los genes involucrados en una enfermedad de este tipo utilizando información de antecedentes familiares. El método GWAS se basa en una base de datos genéticos que ha estado en desarrollo desde 2002 llamada Proyecto Internacional HapMap. El Proyecto HapMap ordenó los genomas de varios cientos de individuos de todo el mundo e identificó grupos de SNP. Los grupos incluyen SNP que se encuentran cerca uno del otro en los cromosomas, por lo que tienden a permanecer juntos a través de la recombinación. El hecho de que el grupo se mantenga unido significa que identificar un marcador SNP es todo lo que se necesita para identificar todos los SNP del grupo. Se identifican varios millones de SNP, pero identificarlos en otras personas a las que no se les ha secuenciado el genoma completo es mucho más fácil porque solo es necesario identificar los SNP marcadores.
En un diseño común para un GWAS, se eligen dos grupos de individuos; un grupo tiene la enfermedad y el otro grupo no. Los individuos en cada grupo se combinan en otras características para reducir el efecto de las variables de confusión que causan diferencias entre los dos grupos. Por ejemplo, los genotipos pueden diferir porque los dos grupos se toman principalmente de diferentes partes del mundo. Una vez que los individuos son elegidos, y típicamente sus números son mil o más para que el estudio funcione, se obtienen muestras de su ADN. El ADN se analiza utilizando sistemas automatizados para identificar grandes diferencias en el porcentaje de SNP particulares entre los dos grupos. A menudo, el estudio examina un millón o más SNP en el ADN. Los resultados de GWAS pueden usarse de dos maneras: las diferencias genéticas pueden usarse como marcadores de susceptibilidad a la enfermedad en individuos no diagnosticados, y los genes particulares identificados pueden ser objetivos para la investigación de la vía molecular de la enfermedad y las posibles terapias. Una consecuencia del descubrimiento de asociaciones genéticas con enfermedades ha sido la formación de compañías que proporcionan la llamada "genómica personal" que identificará los niveles de riesgo para diversas enfermedades en función del complemento SNP de un individuo. La ciencia detrás de estos servicios es controvertida.
Debido a que GWAS busca asociaciones entre genes y enfermedades, estos estudios proporcionan datos para otras investigaciones sobre causas, en lugar de responder preguntas específicas ellos mismos. Una asociación entre una diferencia genética y una enfermedad no significa necesariamente que exista una relación de causa y efecto. Sin embargo, algunos estudios han proporcionado información útil sobre las causas genéticas de las enfermedades. Por ejemplo, tres estudios diferentes en 2005 identificaron un gen para una proteína involucrada en la regulación de la inflamación en el cuerpo que está asociada con una ceguera que causa la enfermedad llamada degeneración macular relacionada con la edad. Esto abrió nuevas posibilidades para la investigación de la causa de esta enfermedad. Se ha identificado que una gran cantidad de genes están asociados con la enfermedad de Crohn usando GWAS, y algunos de ellos han sugerido nuevos mecanismos hipotéticos para la causa de la enfermedad.
Farmacogenómica
La farmacogenómica implica evaluar la efectividad y la seguridad de los medicamentos sobre la base de la información de la secuencia genómica de un individuo. La información personal de la secuencia del genoma se puede usar para prescribir medicamentos que serán más efectivos y menos tóxicos en función del genotipo del paciente individual. Estudiar los cambios en la expresión génica podría proporcionar información sobre el perfil de transcripción génica en presencia del medicamento, que puede usarse como un indicador temprano del potencial de efectos tóxicos. Por ejemplo, los genes involucrados en el crecimiento celular y la muerte celular controlada, cuando se alteran, podrían conducir al crecimiento de células cancerosas. Los estudios de todo el genoma también pueden ayudar a encontrar nuevos genes implicados en la toxicidad de los medicamentos. Las firmas genéticas pueden no ser completamente precisas, pero pueden analizarse más antes de que surjan síntomas patológicos.
 |
| La metagenómica implica aislar el ADN de múltiples especies dentro de un nicho ambiental. El ADN se corta y secuencia, lo que permite que se reconstruyan secuencias completas del genoma de múltiples especies a partir de las secuencias de piezas superpuestas. |
Creación de nuevos biocombustibles
El conocimiento de la genómica de los microorganismos se está utilizando para encontrar mejores formas de aprovechar los biocombustibles de algas y cianobacterias. Las principales fuentes de combustible hoy en día son el carbón, el petróleo, la madera y otros productos vegetales, como el etanol. Aunque las plantas son recursos renovables, aún es necesario encontrar más fuentes alternativas de energía renovable para satisfacer las demandas energéticas de nuestra población. El mundo microbiano es uno de los mayores recursos para genes que codifican nuevas enzimas y producen nuevos compuestos orgánicos, y permanece en gran medida sin explotar. Este vasto recurso genético tiene el potencial de proporcionar nuevas fuentes de biocombustibles.
Genómica mitocondrial
Las mitocondrias son orgánulos intracelulares que contienen su propio ADN. El ADN mitocondrial muta a un ritmo rápido y a menudo se usa para estudiar las relaciones evolutivas. Otra característica que hace interesante el estudio del genoma mitocondrial es que en la mayoría de los organismos multicelulares, el ADN mitocondrial se transmite de la madre durante el proceso de fertilización. Por esta razón, la genómica mitocondrial a menudo se usa para rastrear la genealogía.
Genómica en análisis forense
La información y las pistas obtenidas de las muestras de ADN encontradas en la escena del crimen se han utilizado como evidencia en casos judiciales, y los marcadores genéticos se han utilizado en el análisis forense. El análisis genómico también se ha vuelto útil en este campo. En 2001, se publicó el primer uso de genómica en medicina forense. Fue un esfuerzo de colaboración entre las instituciones de investigación académica y el FBI para resolver los misteriosos casos de ántrax transportados por el Servicio Postal de los EE. UU. La bacteria del ántrax se convirtió en un polvo infeccioso y se envió por correo a los medios de comunicación y a dos senadores estadounidenses. El polvo infectó al personal administrativo y a los trabajadores postales que abrieron o manejaron las cartas. Cinco personas murieron y 17 se enfermaron por la bacteria. Utilizando la genómica microbiana, los investigadores determinaron que se usaba una cepa específica de ántrax en todos los envíos; finalmente, la fuente fue rastreada por un científico en un laboratorio nacional de biodefensa en Maryland.
| Bacillus anthracis es el organismo que causa el ántrax. |
Genómica en agricultura
La genómica puede reducir los ensayos y fracasos involucrados en la investigación científica hasta cierto punto, lo que podría mejorar la calidad y la cantidad de los rendimientos de los cultivos en la agricultura. Vincular los rasgos con los genes o las firmas genéticas ayuda a mejorar la mejora de los cultivos para generar híbridos con las cualidades más deseables. Los científicos usan datos genómicos para identificar rasgos deseables, y luego transfieren esos rasgos a un organismo diferente para crear un nuevo organismo genéticamente modificado, como se describe en el módulo anterior. Los científicos están descubriendo cómo la genómica puede mejorar la calidad y cantidad de la producción agrícola. Por ejemplo, los científicos podrían usar rasgos deseables para crear un producto útil o mejorar un producto existente, como hacer que un cultivo sensible a la sequía sea más tolerante a la estación seca.
| Las plantas agrícolas transgénicas se pueden hacer para resistir enfermedades. Estas ciruelas transgénicas son resistentes al virus de la viruela del ciruelo. |
Las proteínas son los productos finales de los genes que realizan la función codificada por el gen. Las proteínas están compuestas de aminoácidos y juegan papeles importantes en la célula. Todas las enzimas (excepto las ribozimas) son proteínas y actúan como catalizadores que afectan la velocidad de las reacciones. Las proteínas también son moléculas reguladoras, y algunas son hormonas. Las proteínas de transporte, como la hemoglobina, ayudan a transportar oxígeno a varios órganos. Los anticuerpos que defienden contra partículas extrañas también son proteínas. En el estado enfermo, la función de la proteína puede verse afectada debido a cambios a nivel genético o por impacto directo sobre una proteína específica.
Un proteoma es el conjunto completo de proteínas producidas por un tipo celular. Los proteomas se pueden estudiar utilizando el conocimiento de los genomas porque los genes codifican los ARNm, y los ARNm codifican proteínas. El estudio de la función de los proteomas se llama proteómica. La proteómica complementa la genómica y es útil cuando los científicos quieren probar sus hipótesis basadas en genes. Aunque todas las células en un organismo multicelular tienen el mismo conjunto de genes, el conjunto de proteínas producidas en diferentes tejidos es diferente y depende de la expresión génica. Por lo tanto, el genoma es constante, pero el proteoma varía y es dinámico dentro de un organismo. Además, los ARN pueden empalmarse alternativamente (cortarse y pegarse para crear nuevas combinaciones y nuevas proteínas), y muchas proteínas se modifican después de la traducción. Aunque el genoma proporciona un plan, la arquitectura final depende de varios factores que pueden cambiar la progresión de los eventos que generan el proteoma.
Se están estudiando genomas y proteomas de pacientes que padecen enfermedades específicas para comprender la base genética de la enfermedad. La enfermedad más prominente que se estudia con enfoques proteómicos es el cáncer. Se están utilizando enfoques proteómicos para mejorar la detección y la detección temprana del cáncer; Esto se logra mediante la identificación de proteínas cuya expresión se ve afectada por el proceso de la enfermedad. Una proteína individual se denomina biomarcador, mientras que un conjunto de proteínas con niveles de expresión alterados se denomina firma proteica. Para que un biomarcador o una firma de proteína sea útil como candidato para la detección temprana y la detección de un cáncer, debe secretarse en fluidos corporales como el sudor, la sangre o la orina, de modo que las evaluaciones a gran escala puedan realizarse de manera no invasiva . El problema actual con el uso de biomarcadores para la detección temprana del cáncer es la alta tasa de resultados falsos negativos. Un resultado falso negativo es un resultado negativo de la prueba que debería haber sido positivo. En otras palabras, muchos casos de cáncer no se detectan, lo que hace que los biomarcadores no sean confiables. Algunos ejemplos de biomarcadores de proteínas utilizados en la detección de cáncer son CA-125 para cáncer de ovario y PSA para cáncer de próstata. Las firmas de proteínas pueden ser más confiables que los biomarcadores para detectar células cancerosas. La proteómica también se está utilizando para desarrollar planes de tratamiento individualizados, lo que implica la predicción de si un individuo responderá o no a medicamentos específicos y los efectos secundarios que el individuo pueda tener. La proteómica también se está utilizando para predecir la posibilidad de recurrencia de la enfermedad.
| Esta máquina se está preparando para hacer un análisis de patrones proteómicos para identificar cánceres específicos para poder hacer un pronóstico preciso del cáncer. |
El National Cancer Institute ha desarrollado programas para mejorar la detección y el tratamiento del cáncer. Las Tecnologías de Proteómica Clínica para el Cáncer y la Red de Investigación de Detección Temprana son esfuerzos para identificar firmas de proteínas específicas para diferentes tipos de cáncer. El Programa de Proteómica Biomédica está diseñado para identificar firmas de proteínas y diseñar terapias efectivas para pacientes con cáncer.
¿Te sirvió el contenido del tema? ¿tienes alguna duda o sugerencia?
Puedes escribir un comentario en la parte de abajo y nosotros con gusto te responderemos. Esperemos que tengas un lindo día. ¡Mucho éxito en tus estudios!
Puedes escribir un comentario en la parte de abajo y nosotros con gusto te responderemos. Esperemos que tengas un lindo día. ¡Mucho éxito en tus estudios!
